¿Cómo se detecta si un paciente está infectado por coronavirus?
Saber detectar el coronavirus es una buena noticia. Cuando la detección sea masiva, será una buenísima noticia
Una de los tecnicismos relacionados con la pandemia actual que ha trascendido al público general es el de “PCR”, ese sistema que de alguna forma permite detectar el coronavirus SARS-CoV-2 [contexto id=”460724″]. Para ser exactos, el método recibe el nombre de RT-PCR en tiempo real (RT-qPCR). Pero, ¿cómo se caza exactamente al virus?
El método proporciona un resultado en 4 o 5 horas. Ese tiempo es posible solo cuando se analizan muestras de un único paciente. Desgraciadamente, el tiempo de respuesta es muy superior. Cientos de pacientes acuden a los hospitales cada día para ser diagnosticados, y los recursos materiales y humanos son insuficientes.
A sus anchas en las vías respiratorias
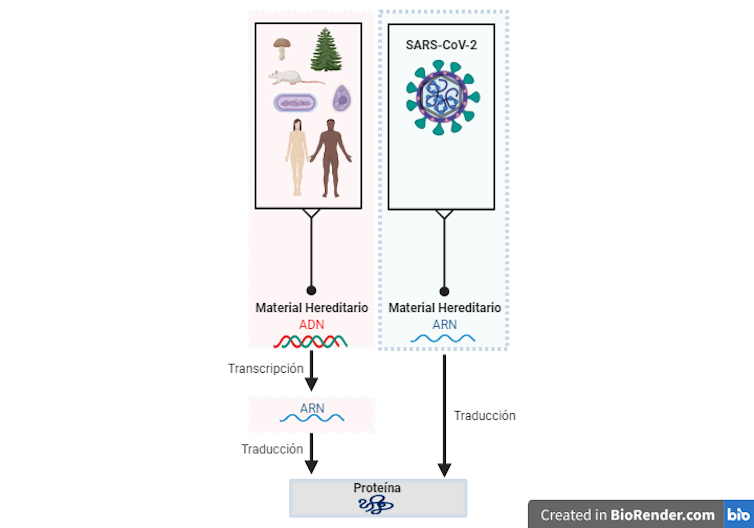
El material genético del SARS-CoV-2 es ARN. Este se suele detectar en muestras del tracto respiratorio superior e inferior de pacientes que, tras una evaluación médica, cumplen los criterios de caso en investigación. Estos son: tener los síntomas principales de la enfermedad, la historia clínica previa y el contacto reciente con contagiados.
Un método molecular de detección rápida
El protocolo de detección comienza tras recibir las muestras respiratorias. La detección es indirecta: lo que se detecta es el ARN del coronavirus.
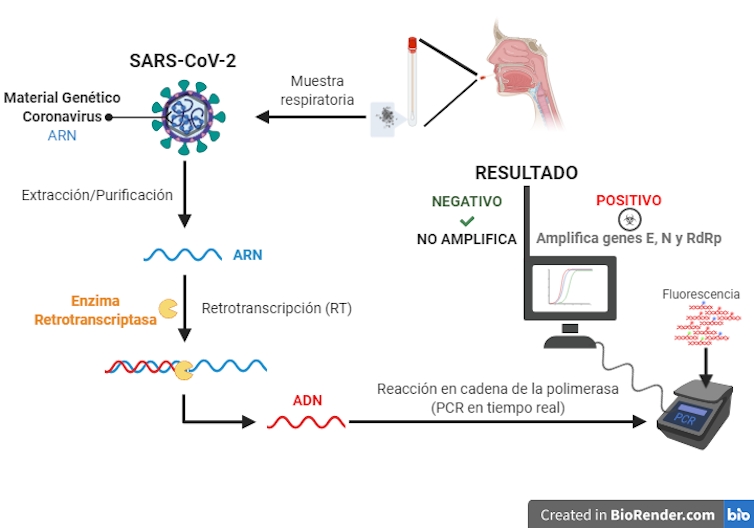
Primeramente se extrae y purifica el ARN. Luego se realiza la reacción RT-qPCR. Son dos reacciones diferentes, pero la RT y la qPCR se llevan a cabo al mismo tiempo en un tubo para reducir tiempo, recursos y manipulaciones innecesarias. Ambas reacciones reproducen in vitro procesos que ocurren in vivo gracias a enzimas especiales.
Veamos cada una de ellas por separado.
RT va de ReTro
La PCR en la que se basa la técnica copia ADN, no ARN. Esto supone un problema para detectar un microorganismo que no tiene ADN pero, por suerte, es de fácil solución. El ARN del virus purificado se convierte en ADN mediante un proceso conocido como retrotranscripción (RT). La enzima encargada es la retrotranscriptasa o transcriptasa inversa.
La retrotranscripción es la forma de multiplicación habitual de los retrovirus. Por eso se usan enzimas purificadas de retrovirus como el de la mieloblastosis aviar y el de la leucemia murina de Moleney.
La PCR es una “fotocopiadora” molecular
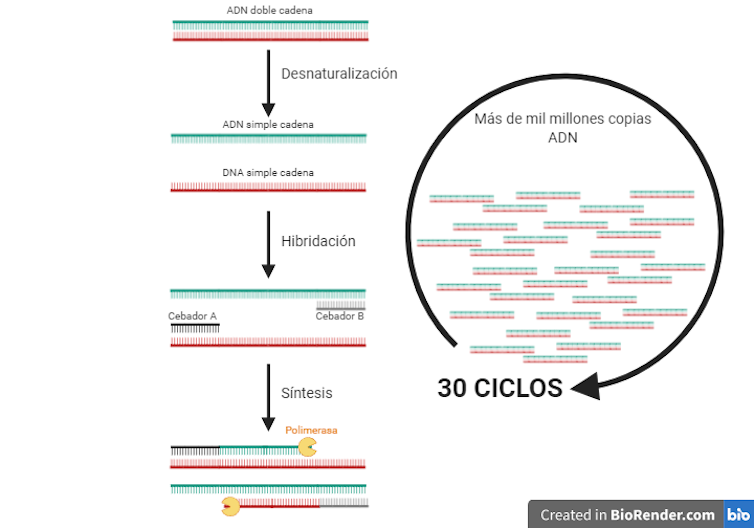
Cuando una célula se divide duplica su ADN con la enzima ADN polimerasa. El proceso se llama síntesis. La PCR imita esta síntesis y la repite cíclicamente hasta conseguir multiplicar (amplificar) el ADN molde presente en la reacción. Por eso PCR se traduce del inglés como “reacción en cadena de la polimerasa”.
La reacción produce en condiciones ideales 2³⁰ moléculas (aproximadamente mil millones) por cada molécula de ADN presente. Las polimerasas usadas son de microorganismos que crecen a temperaturas extremadamente altas. La Taq polimerasa de la bacteria Thermus aquaticus es una de ellas.
La PCR no amplifica todo el material genético del virus, solo trozos de genes. Concretamente el E, el N y el RdRp. E codifica una de las proteínas de la envuelta del virus. N codifica la proteína de la nucleocápsida o armazón proteico del virus. RdRp, la enzima que copia el ARN del virus, llamada transcriptasa dependiente de ARN.
La secuencia genómica del SARS-CoV-2 y la de los de otros coronavirus son parecidas, pero no idénticas. La PCR aprovecha las diferencias en los genes mencionados para discriminar entre virus. Para ello usa unos cebadores altamente específicos que contienen las diferencias en sus secuencias.
Un cebador no es más que una pequeña molécula de ADN necesaria para iniciar el proceso de síntesis. Si un cebador no se une perfectamente a su molde de ADN, la polimerasa no es capaz de iniciar la síntesis. Eso es lo que ocurre cuando los cebadores intentan amplificar ADN que no pertenece al SARS-CoV-2. No se unen al molde y no amplifican.
Tres claves para la detección
Tener una fotocopiadora molecular de ADN es la primera de las claves. Detectar una única molécula es casi imposible, detectar miles de millones de copias es sencillo.
Las otras claves son la alta sensibilidad y especificidad de la PCR. Ambas son necesarias para detectar inequívocamente el ARN del SARS-CoV-2 y no el de otros virus ni el del paciente.
La sensibilidad informa de la presencia de falsos negativos. Si no los hay, la sensibilidad es 100 %. En plena expansión de contagios, es importantísimo no tener falsos negativos. Es tranquilizador, por tanto, que la sensibilidad del método esté por encima del 95 %.
La especificidad informa de la presencia de falsos positivos. La especificidad es 100 % si no los hay. Altas sensibilidad y especificidad se consiguen gracias a los cebadores específicos mencionados arriba.
Lo ideal es que la PCR detecte el 100 % de los verdaderos positivos y el 0 % de los verdaderos negativos.
La tecnología tiempo real mide fluorescencia
Hasta ahora tenemos que el ARN se extrae y se purifica de las muestras respiratorias, se convierte en ADN, que se multiplica para producir miles de millones de copias de los genes E, N y RdRp. Esos millones de copias se observan gracias a un compuesto fluorescente que se une al ADN. A mayor número de copias de ADN, mayor fluorescencia.
La fluorescencia, acumulada a medida que transcurren los ciclos de PCR, se registra en tiempo real con un termociclador. Es el aparato donde se incuba la reacción RT-qPCR. La letra q (del inglés quantitative), obviada hasta ahora, hace referencia a que el valor de fluorescencia es cuantificable.
Terminada la qPCR y analizados los resultados, una muestra se considera positiva por coronavirus cuando los valores de fluorescencia de los tres genes (E, N y RdRp) están por encima de un umbral preestablecido.
Saber detectar el coronavirus es una buena noticia. Cuando la detección sea masiva, será una buenísima noticia.![]()
Este artículo fue publicado originalmente en The Conversation. Lea el original.
![]()

